Durante décadas, prever como uma proteína se dobra no espaço foi um dos maiores desafios da ciência. Cada sequência de aminoácidos pode assumir formas tridimensionais complexas e, até pouco tempo atrás, só era possível descobrir essas formas por meio de experimentos caros e demorados.
Neste episódio, Bruno Franchi e Amira Al Dergham contam como a inteligência artificial mudou esse jogo. Da cristalografia de raios-X às competições do CASP, passando por gamers que ajudaram na pesquisa, você vai conhecer o caminho que levou ao AlphaFold, o sistema da DeepMind que previu a estrutura de mais de 200 milhões de proteínas com precisão inédita, um feito que rendeu o Nobel de Química em 2024.
Uma história que une biologia, química, física e IA para resolver um problema que intrigou gerações de cientistas.
Este episódio contou com o apoio da FAPESP e da Fundação Roberto Marinho.
REFERÊNCIAS
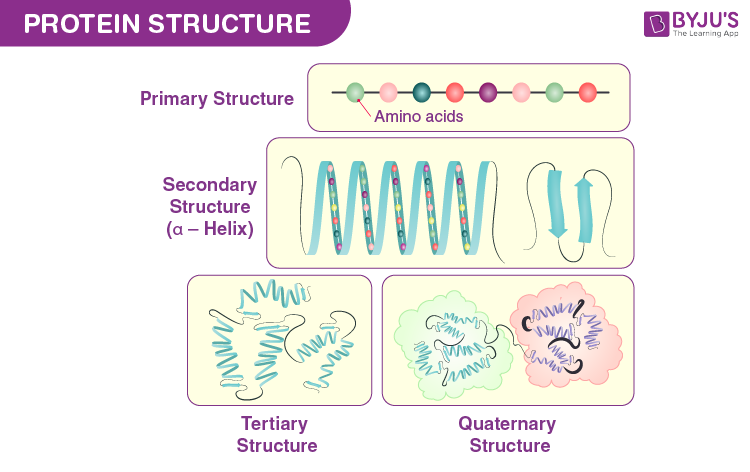
ESTRUTURA DA PROTEÍNA
PRÊMIOS NOBEL E FIGURAS HISTÓRICAS
- Max von Laue (1914) – Nobel de Física pela descoberta de que raios-X poderiam gerar padrões de difração em cristais.
- William Henry Bragg e William Lawrence Bragg (1915) – Nobel de Física pelo desenvolvimento da cristalografia de raios-X.
- Max Perutz e John Kendrew (1962) – Nobel de Química pelos estudos da estrutura da hemoglobina e mioglobina.
- Christian Anfinsen (1972) – Nobel de Química pela demonstração de que a informação para o enovelamento está na sequência primária de aminoácidos.
- Nobel Prize em química de 2024 – Premiação pelo desenvolvimento do AlphaFold e pela revolução na previsão de estruturas proteicas.
MARCOS E INSTITUIÇÕES CIENTÍFICAS
- Protein Data Bank (PDB) – Repositório global de acesso aberto que armazena dados tridimensionais de macromoléculas biológicas.
- Sírius – Fonte de luz síncrotron de 4ª geração, localizada em Campinas, utilizada para estudos avançados de cristalografia.
- CASP (Critical Assessment of Protein Structure Prediction) – Competição bienal para avaliar métodos de previsão de estruturas de proteínas.
TECNOLOGIAS E PROJETOS
- Cristalografia de raio-X – Técnica para determinar estruturas tridimensionais de proteínas e outras moléculas cristalinas.
- Rosetta – Algoritmo criado por David Baker para prever estruturas de proteínas.
- FoldIt – Jogo colaborativo em que jogadores ajudaram a resolver a estrutura de uma proteína do HIV, publicado no artigo “Crystal Structure of Monomeric Retroviral Protease solved by protein folding game players”.
- AlphaGo – Algoritmo de IA da DeepMind que venceu o campeão mundial de Go, Lee Sedol, em 2016.
- AlphaFold – Algoritmo de IA da DeepMind capaz de prever a estrutura tridimensional de proteínas com alta precisão.
PERSONAGENS
- David Baker – Criador do algoritmo Rosetta e do projeto FoldIt.
- Demis Hassabis – Fundador da DeepMind e criador dos algoritmos AlphaGo e AlphaFold.
CURIOSIDADES E MENÇÕES NA MÍDIA
- The Guardian (2013) – Artigo apelidando a maquete da estrutura da mioglobina de “o cocô do século”.
APRESENTAÇÃO
Amira Romagnoli Al Dergham | Midialogia – Unicamp
Bruno Franchi | Física – Unicamp
ROTEIRO:
Bruno Franchi
Heitor Fasina
REVISÃO:
Victor Vicente
Leandro Tessler
COORDENAÇÃO GERAL:
Leandro Tessler
Victor Vicente
EDIÇÃO:
Aluísio Augusto
APOIO:
FAPESP [Bolsa Comunicar Ciência]
Fundação Roberto Marinho
REALIZAÇÃO:
BI0S – Brazilian Institute of Data Science | Unicamp